FDT - Pré-processamento
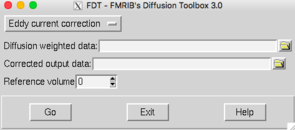
Eddy current correction:
- Input: DWI.nii.gz (Arquivo de difusão da ressonância em nifti)
- Ref Volume: 0
- GO
- Output: data.nii.gz (corrigida)
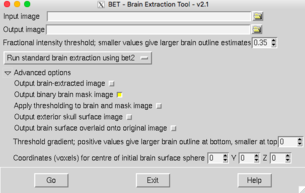
Bet brain extraction:
Criação de mask binária
- Input: data.nii.gz (corrigida para eddy)
- Threshold = 0.35
- Advanced options > Output binary brain mask image
- GO
- Output: data_brain_mask.nii.gz
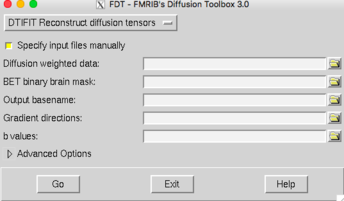
DIFTI:
Fazer inputs manualmente:
- data.nii.gz (corrigida)
- data_brain_mask.nii.gz (mask)
- arquivo.bvec
- arquivo.bval
- GO
Outputs:
dti_FA.nii.gz ; dti_MD.nii.gz ; dti_MO.nii.gz ; dti_V1.nii.gz ; dti_V2.nii.gz ; dti_V3.nii.gz ; dti_L1.nii.gz ; dti_L2.nii.gz ; dti_L3.nii.gz ; dti_S0.nii.gz
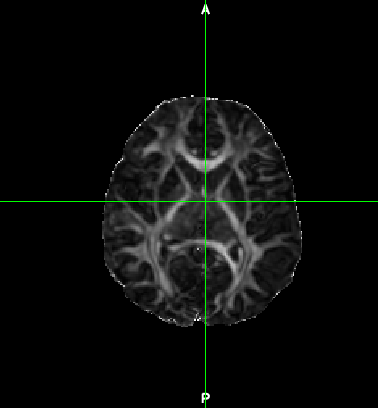
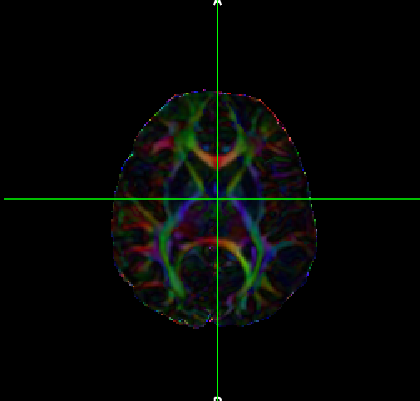
Visualização dos mapas de FA (com ou sem modulação por V1) via terminal:
| cd [caminho_diretorio_subj] fsleyes dti_FA.nii.gz |
| #! /bin/bash
# Realiza o pré-processamento dos arquivos de DWI até o cálculo dos mapas de FA # Inclui: EDDY CURRENT CORRECTION ; BRAIN MASK EXTRACTION ; FITTING TENSORS subj = 01 pasta = /caminho_até_diretório/DTI_${subj} cd ${pasta} eddy_correct dwi.nii.gz dwic.nii.gz 0 bet dwic.nii.gz dti_brain.nii.gz -f 0.35 -m dtifit -k dwic.nii.gz -m dti_brain.nii.gz -r ${subj}.bvec -b ${subj}.bval cp ${pasta}/${subj}_FA.nii.gz /caminho_até_diretório_para_análise/ [OPCIONAL] echo done |
Basta configurar o script para seus diretórios e nomes de sujeitos. O arquivo dwi.nii.gz e os arquivos .bvec e .bval devem ter seus nomes alterados conforme o script ou a preferência.
TBSS - Processamento:
1 - Criar uma pasta onde será realizado o TBSS e copiar os arquivos de dti_FA.nii.gz dos sujeitos para a pasta.
2 - Trocar os nomes dos arquivos pela identificação dos sujeitos - na hora em que serão extraídos os valores de FA eles estarão nesta ordem.
Na pasta com os arquivos de FA, realizar os 3 primeiros passos de processamento, da seguinte forma:
| tbss_1_preproc *.nii.gz
tbss_2_reg -T tbss_3_postreg -S cd stats fsleyes -std1mm mean_FA -cm red-yellow -dr 0.2 0.6 & |
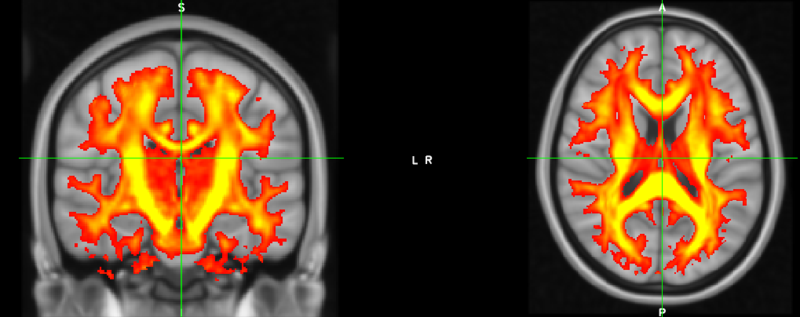
Processamento:
- Remoção de alguns artefatos
- Criação de uma pasta FA (novos arquivos processados) e origdata (arquivos originais)
- Alinhamento de todos as FA em um template do FSL chamado FMRIB58_FA
- Fusão e registro das FA em um espaço padrão 1x1x1mm - Criação de um esqueleto com as FA
O processamento inicial tem como outputs principais:
mean_FA_skeleton.nii.gz
mean_FA.nii.gz
mean_FA_mask.nii.gz
all_FA.nii.gz
Voltar para a pasta tbss e rodar o último processamento
| cd ..
tbss_4_prestats 0.3 |
O último comando determina o threshold para mean_FA e projeta os mapas em um arquivo de esqueleto 4D que alimentará a análise estatística.
Output: all_FA_skeletonised.nii.gz
TBSS - General Linear Model (Análise):
Entrar no diretório de stats e iniciar a ferramenta estatística:
| cd stats
Glm |
Aqui deve-se selecionar o número de sujeitos que entrarão no modelo (inputs) NA ORDEM ALFABÉTICA QUE FORAM PROCESSADOS. Na interface do GLM clicar em Wizard e desenhar o modelo que deseja-se trabalhar (número de contrastes, grupo, ...). Salvar o modelo no diretório stats como design
Rodar o TBSS randomise
| randomise -i all_FA_skeletonised -o tbss \
-m mean_FA_skeleton_mask -d design.mat -t design.con -c 1.5 |
| randomise -i all_FA_skeletonised -o tbss \
-m mean_FA_skeleton_mask -d design.mat -t design.con --T2 |
Outputs:
- tbss_tstat1 (arquivo cru, sem thresholds)
- tbss_clustere_corrp_tstat1 (cluster com os p-values corrigidos para múltiplas comparações)
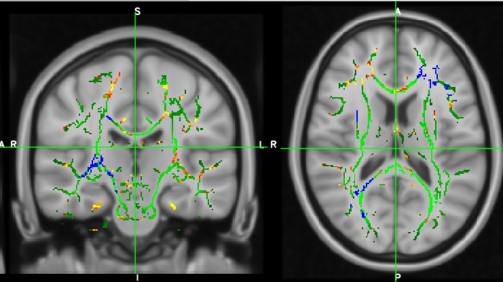
Visualizar os resultados em um threshold para p=0.05
| fsleyes -std1mm mean_FA_skeleton -cm green -dr .3 .7 \
tbss_tstat1 -cm red-yellow -dr 1.5 3 \ tbss_clustere_corrp_tstat1 -cm blue-lightblue -dr 0.949 1 & |
Em azul - tractos que atingiram threshold (p=0.05)
Para uma visualização mais "bonita", rodar o seguinte script:
| tbss_fill tbss_clustere_corrp_tstat1 0.95 \
mean_FA tbss_clustere_corrp_tstat1_filled fsleyes -std1mm mean_FA_skeleton -cm green -dr .3 .7 \ tbss_clustere_corrp_tstat1_filled.nii.gz -cm red-yellow |
Os tractos que atingirem o threshold (p=0.05) ficarão em vermelho e mais "cheios".
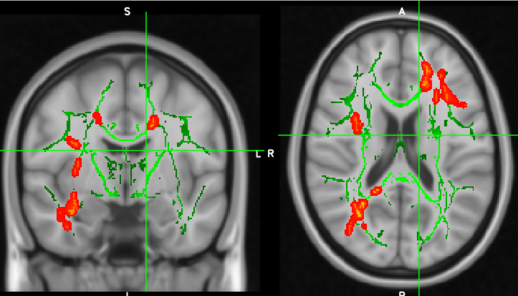
Pode-se utilizar algum atlas para identificar os tractos estatisticamente significativos.
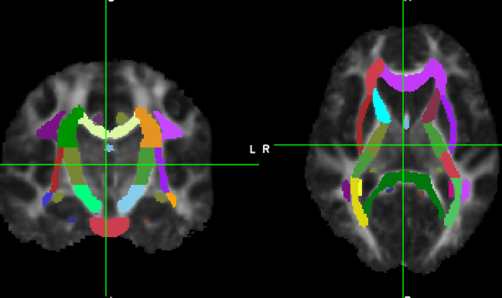
Análise de Regiões de Interesse (ROIs) e extração de valores de Fração de Anisotropia (FA)
#Para esta parte do guia utilizaremos o atlas de DTI da Johns Hopkins University (JHU) e a ferramenta "fslmeants" do FSL
1. Publicação do Atlas: Link
O atlas que vem com o FSL chamado JHU-ICBM-labels-1mm.nii.gz descreve 48 ROIs em substância branca e define coordenadas de probabilidade onde passam os tractos de interesse no espaço MNI152.
Podemos criar máscaras utilizando fslmaths e as coordenadas já pré definidas por este atlas e extrair valores médios de FA utilizando essas máscaras, correspondendo aos valores médios de FA nos tractos de interesse de cada estudo.
As ROIs e seus respectivos índices dentro do atlas são as seguintes:
- 1>Cerebellar Penducle
- 2>Pontine crossing tract (a part of MCP)
- 3>Genu of corpus callosum
- 4>Body of corpus callosum
- 5>Splenium of corpus callosum
- 6>Fornix (column and body of fornix)
- 7>Corticospinal tract R
- 8>Corticospinal tract L
- 9>Medial lemniscus R
- 10>Medial lemniscus L
- 11>Inferior cerebellar peduncle R
- 12>Inferior cerebellar peduncle L
- 13>Superior cerebellar peduncle R
- 14>Superior cerebellar peduncle L
- 15>Cerebral peduncle R
- 16>Cerebral peduncle L
- 17>Anterior limb of internal capsule R
- 18>Anterior limb of internal capsule L
- 19>Posterior limb of internal capsule R
- 20>Posterior limb of internal capsule L
- 21>Retrolenticular part of internal capsule R
- 22>Retrolenticular part of internal capsule L
- 23>Anterior corona radiata R
- 24>Anterior corona radiata L
- 25>Superior corona radiata R
- 26>Superior corona radiata L
- 27>Posterior corona radiata R
- 28>Posterior corona radiata L
- 29>Posterior thalamic radiation (include optic radiation) R
- 30>Posterior thalamic radiation (include optic radiation) L
- 31>Sagittal stratum (include inferior longitidinal fasciculus and inferior fronto-occipital fasciculus) R
- 32>Sagittal stratum (include inferior longitidinal fasciculus and inferior fronto-occipital fasciculus) L
- 33>External capsule R
- 34>External capsule L
- 35>Cingulum (cingulate gyrus) R
- 36>Cingulum (cingulate gyrus) L
- 37>Cingulum (hippocampus) R
- 38>Cingulum (hippocampus) L
- 39>Fornix (cres) / Stria terminalis (can not be resolved with current resolution) R
- 40>Fornix (cres) / Stria terminalis (can not be resolved with current resolution) L
- 41>Superior longitudinal fasciculus R
- 42>Superior longitudinal fasciculus L
- 43>Superior fronto-occipital fasciculus (could be a part of anterior internal capsule) R
- 44>Superior fronto-occipital fasciculus (could be a part of anterior internal capsule) L
- 45>Uncinate fasciculus R
- 46>Uncinate fasciculus L
- 47>Tapetum R
- 48>Tapetum L
Para extrair os valores basta utilizar o seguinte script dentro da pasta do stats:
| #! /bin/bash
#Extração de valores de FA para ROIs #Colocar os roinum conforme o atlas JHU para ROIs dir=[caminho_diretório_stats] cd $dir for roinum in 5 6 7 8 9 10 12 ; do fslmaths /usr/local/fsl/data/atlases/JHU/JHU-ICBM-labels-1mm.nii.gz -thr $roinum -uthr $roinum -bin roimask fslmaths roimask -mas mean_FA_skeleton_mask.nii.gz -bin roimask padroi=`$FSLDIR/bin/zeropad $roinum 3` fslmeants -i all_FA_skeletonised.nii.gz -m roimask -o meants_roi${padroi}.txt done paste meants_roi*.txt > allmeants_roi.txt |
Sendo que na primeira linha deve-se colocar os índices das ROIs que deseja-se extrair. Lembrando que os valores virão conforme a ordem alfabética em que foram processados na etapa inicial do TBSS.
JHU-ICBM-labels-1mm - Atlas
FIM.
Boas análises!
Referências: UserGuide S.M. Smith, M. Jenkinson, H. Johansen-Berg, D. Rueckert, T.E. Nichols, C.E. Mackay, K.E. Watkins, O. Ciccarelli, M.Z. Cader, P.M. Matthews, and T.E.J. Behrens. Tract-based spatial statistics: Voxelwise analysis of multi-subject diffusion data. NeuroImage, 31:1487-1505, 2006.
Dúvidas ou sugestões: rafael.sommer@acad.pucrs.br

